Abbildungen II
Gruppierte Darstellungen
Balkendiagramme
Bei dem Versuch zwei kategoriale mit einer numerischen zu kombinieren oder kombinierte Häufigkeiten von zwei Gruppierungsfaktoren darzustellen kommen wir mit den bisher benutzten aesthetics nicht unbedingt weiter.
Als Beispiel betrachten wir folgenden Ausschnitt des hier verfügbaren Datensatz:
## # A tibble: 21 × 3
## sex age DE
## <chr> <chr> <dbl>
## 1 F Y15-19 82.7
## 2 F Y20-24 73.4
## 3 F Y25-29 73.2
## 4 F Y30-44 76.9
## 5 F Y45-54 74
## 6 F Y55-64 81.7
## 7 F Y65-74 91.1
## 8 M Y15-19 81.4
## 9 M Y20-24 68.7
## 10 M Y25-29 65.1
## # … with 11 more rowsDiese Anteile an Rauchern pro Altersgruppe wollen wir jetzt in einem Balkendiagramm darstellen, in dem an der x-Achse die Altersgruppen abgetragen sind. Außerdem wollen wir die Balken nach Geschlecht gruppieren:
df %>%
ggplot(aes(x = age, fill = sex, y = DE)) +
geom_col()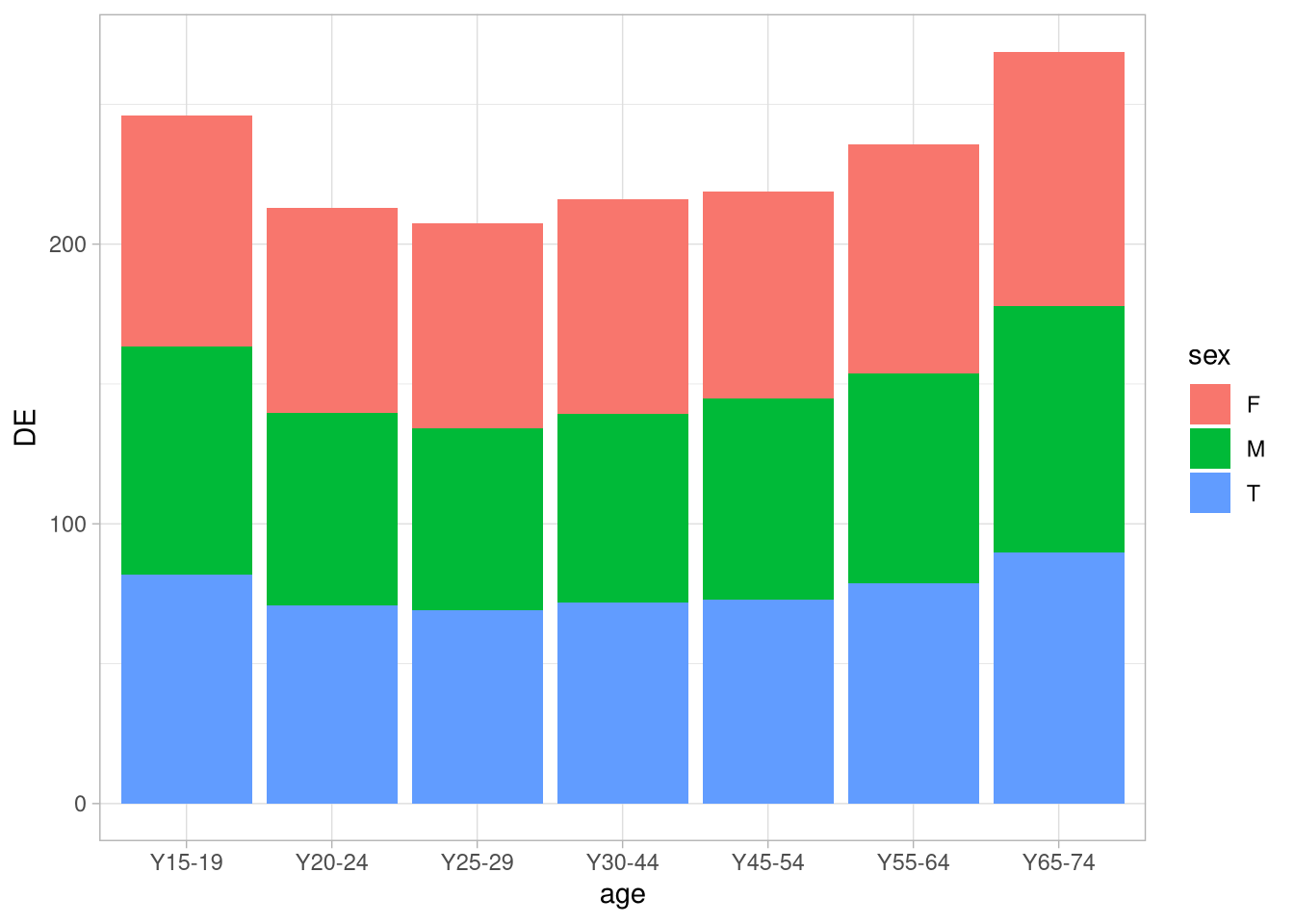 Diese Darstellung ist zwar ganz nett um einen Eindruck für den den Verlauf über die Kohorten zu sehen, die Balken nebeneinander zu gruppieren ist aber vielleicht eher wünschenswert. Dafür können wir das
Diese Darstellung ist zwar ganz nett um einen Eindruck für den den Verlauf über die Kohorten zu sehen, die Balken nebeneinander zu gruppieren ist aber vielleicht eher wünschenswert. Dafür können wir das position-Argument und die position_dodge()-Funktion benutzen:
df %>%
ggplot(aes(x = age, fill = sex, y = DE)) +
geom_col(position = position_dodge())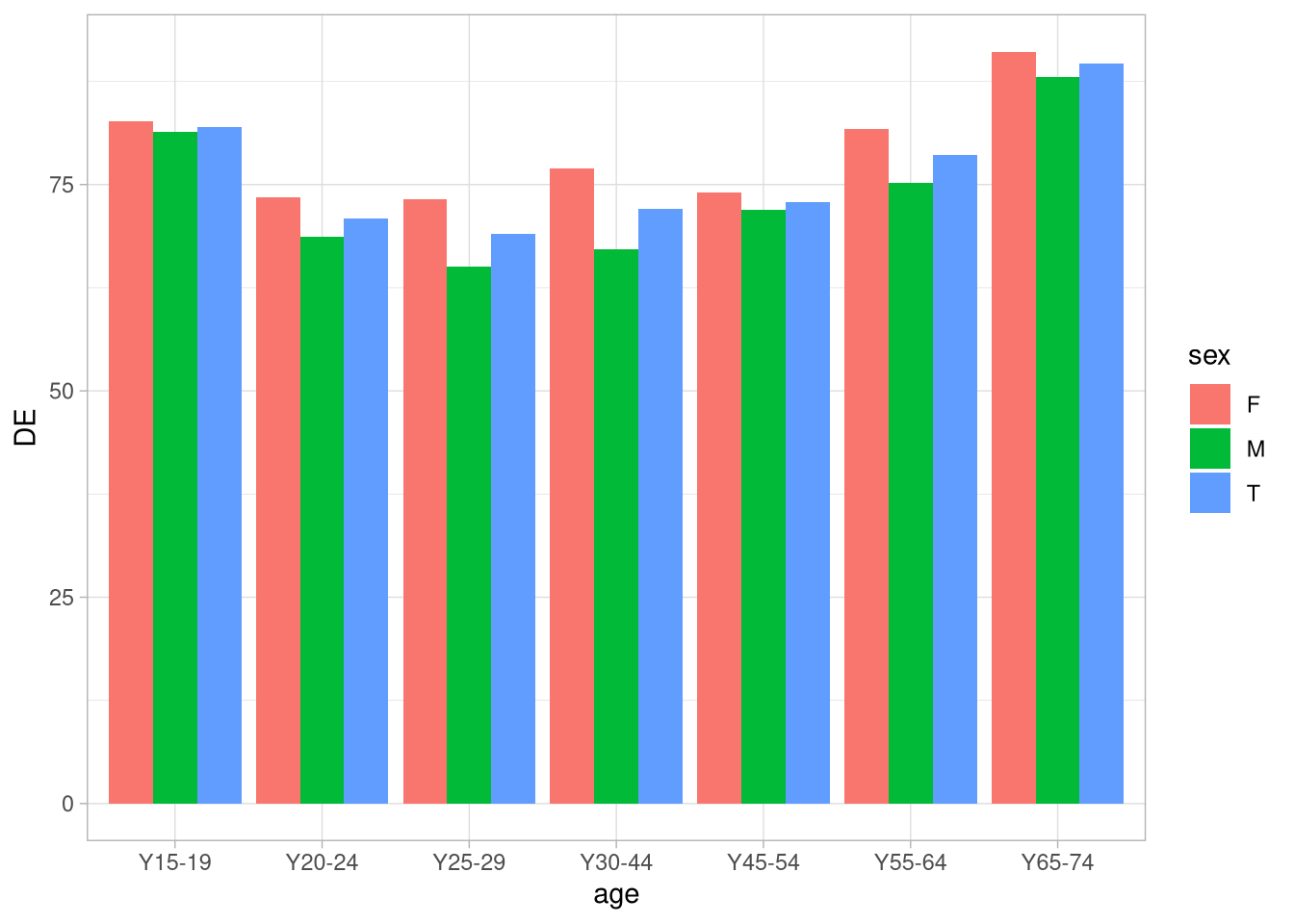
Den Graphen können wir jetzt wie gewohnt verschönern, zum Beispiel indem wir die Farben ändern und die Achsenbeschriftung anpassen:
df %>%
ggplot(aes(x = age, fill = sex, y = DE)) +
geom_col(position = position_dodge()) +
scale_fill_manual(values = c(F = 'darkblue',
M = 'brown3',
T = 'darkorchid4')) +
labs(x = 'Alter',
y = 'Prozent Raucher',
fill = 'Geschlecht',
title = 'Anteil Raucher',
subtitle = 'Pro Altergruppe in Deutschland im Jahr 2014')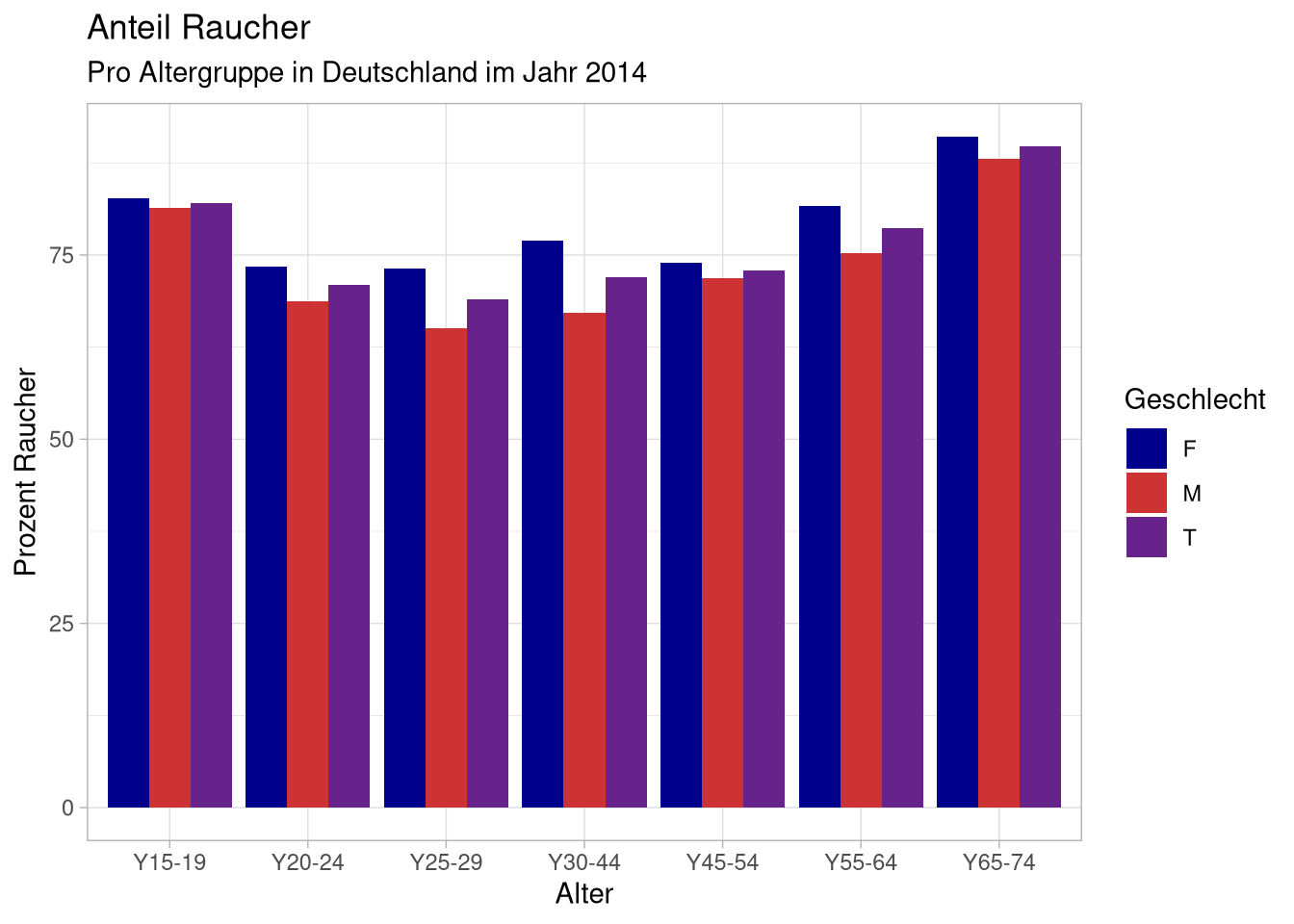
Boxplots
Wenn wir den Datensatz ein bisschen anders aufbereiten, können wir die Verteilungen der Raucher-Anteile über Europa darstellen.
## # A tibble: 651 × 4
## sex age country percentage
## <chr> <chr> <chr> <dbl>
## 1 F Y15-19 BE 76.6
## 2 F Y15-19 BG 83.2
## 3 F Y15-19 CZ 83.5
## 4 F Y15-19 DK 82.1
## 5 F Y15-19 DE 82.7
## 6 F Y15-19 EE 91.2
## 7 F Y15-19 IE 86.3
## 8 F Y15-19 EL 87.7
## 9 F Y15-19 ES 89.8
## 10 F Y15-19 FR 80.8
## # … with 641 more rowsUm einen Eindruck über die Verteilung der Raucherzahlen über Europa zu bekommen, stellen wir diesen Datensatz als gruppierte Boxplots dar:
df %>%
ggplot(aes(x = age,
y = percentage,
color = sex)) +
geom_boxplot()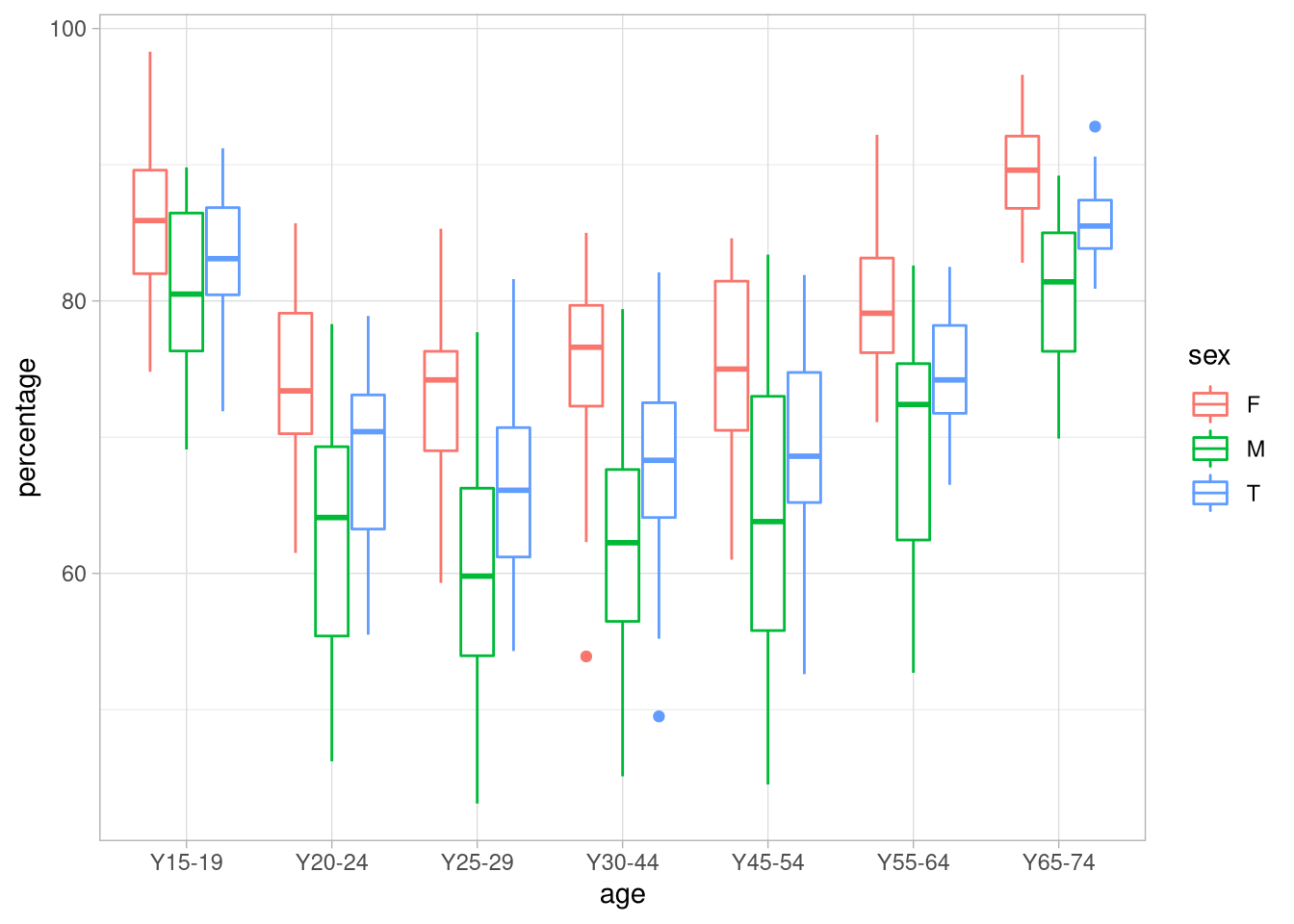 Hier können wir auch wieder die grafischen Optimierungen anwenden, die wir beim Balkendiagramm genutzt haben:
Hier können wir auch wieder die grafischen Optimierungen anwenden, die wir beim Balkendiagramm genutzt haben:
df %>%
ggplot(aes(x = age,
y = percentage,
color = sex)) +
geom_boxplot() +
scale_color_manual(values = c(F = 'darkblue',
M = 'brown3',
T = 'darkorchid4')) +
labs(x = 'Alter',
y = 'Prozent Raucher',
color = 'Geschlecht',
title = 'Anteil Raucher',
subtitle = 'Pro Altergruppe in Europa im Jahr 2014')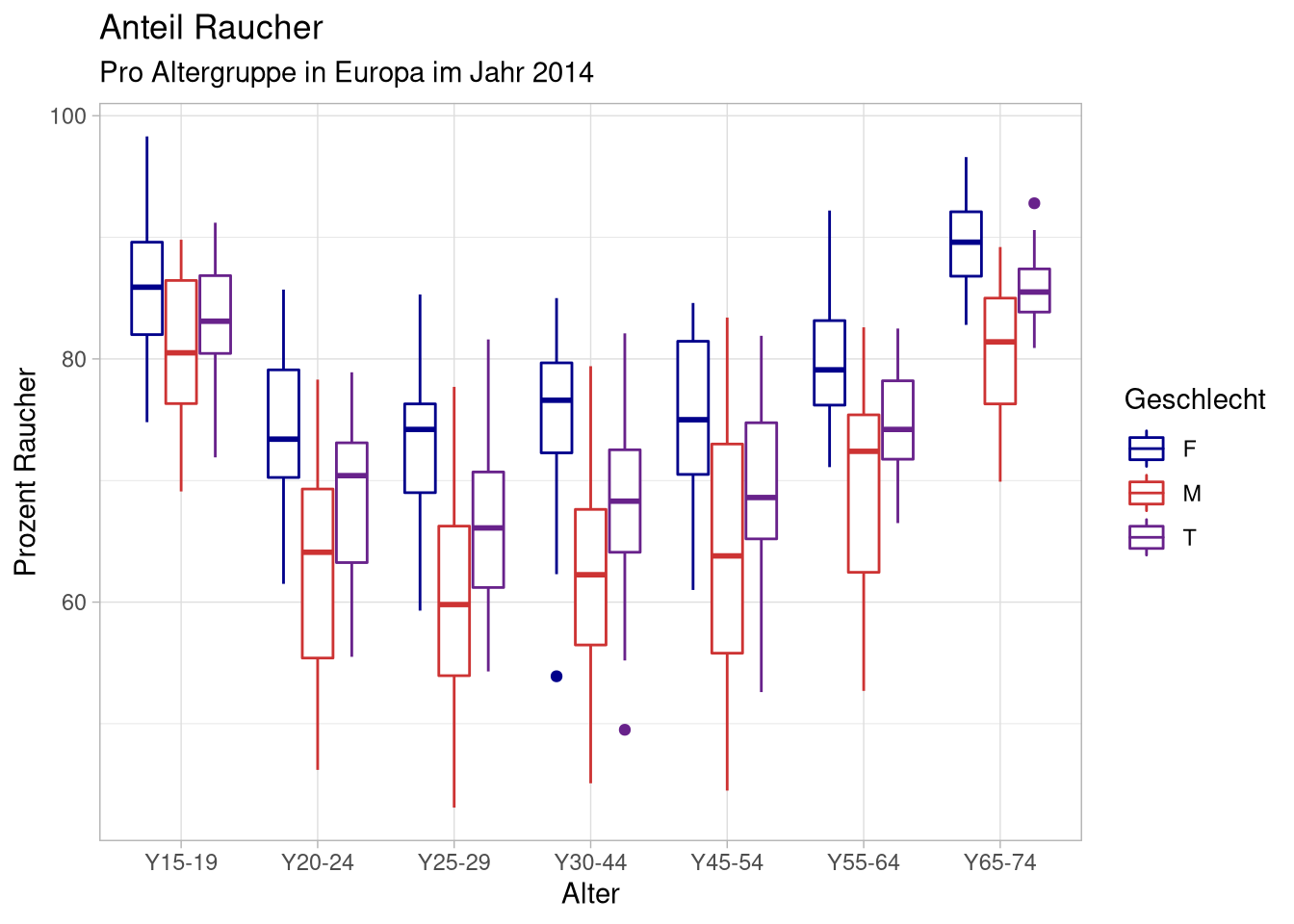
Layer für zusätzliche Informationen
Gruppenzusammenfassungen
Zu unserem gerade erstellten Boxplot wollen wir jetzt die mittleren Raucherquoten als horizontale Linie einfügen.
Dazu erstellen wir zuerst einen Datensatz mit den Mittelwerten:
mean_per_sex <- df %>%
group_by(sex) %>%
summarise(percentage = mean(percentage,na.rm = T))
mean_per_sex## # A tibble: 3 × 2
## sex percentage
## <chr> <dbl>
## 1 F 79.0
## 2 M 68.5
## 3 T 73.9Diese können wir dann als eigenen Datensatz einem neuen geom hinzufügen. Da wir horizontale Linien hinzufügen wollen, nutzen wir dafür geom_hline.
df %>%
ggplot(aes(x = age,
y = percentage,
color = sex)) +
geom_boxplot() +
scale_color_manual(values = c(F = 'darkblue',
M = 'brown3',
T = 'darkorchid4')) +
labs(x = 'Alter',
y = 'Prozent Raucher',
color = 'Geschlecht',
title = 'Anteil Raucher',
subtitle = 'Pro Altergruppe in Europa im Jahr 2014') +
geom_hline(data = mean_per_sex,
mapping = aes(yintercept = percentage,
color = sex))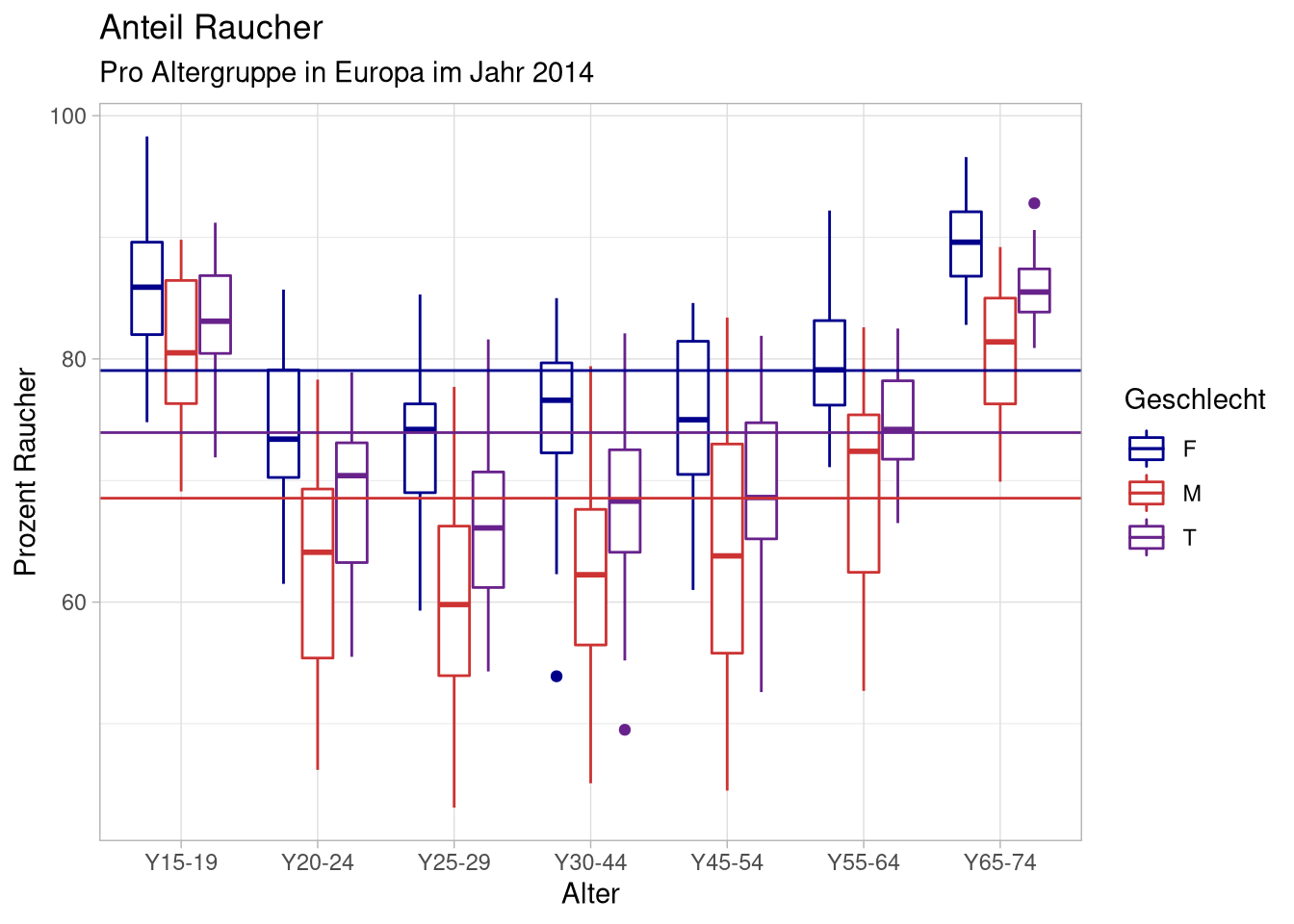
Mit außerhalb der aes-Funktion gesetzten ‘aesthetics’ können wir die Linien noch ein bisschen mehr von den Boxplots abheben.
df %>%
ggplot(aes(x = age,
y = percentage,
color = sex)) +
geom_boxplot() +
scale_color_manual(values = c(F = 'darkblue',
M = 'brown3',
T = 'darkorchid4')) +
labs(x = 'Alter',
y = 'Prozent Raucher',
color = 'Geschlecht',
title = 'Anteil Raucher',
subtitle = 'Pro Altergruppe in Europa im Jahr 2014') +
geom_hline(data = mean_per_sex,
mapping = aes(yintercept = percentage,
color = sex),
alpha = .5,
lty = 2)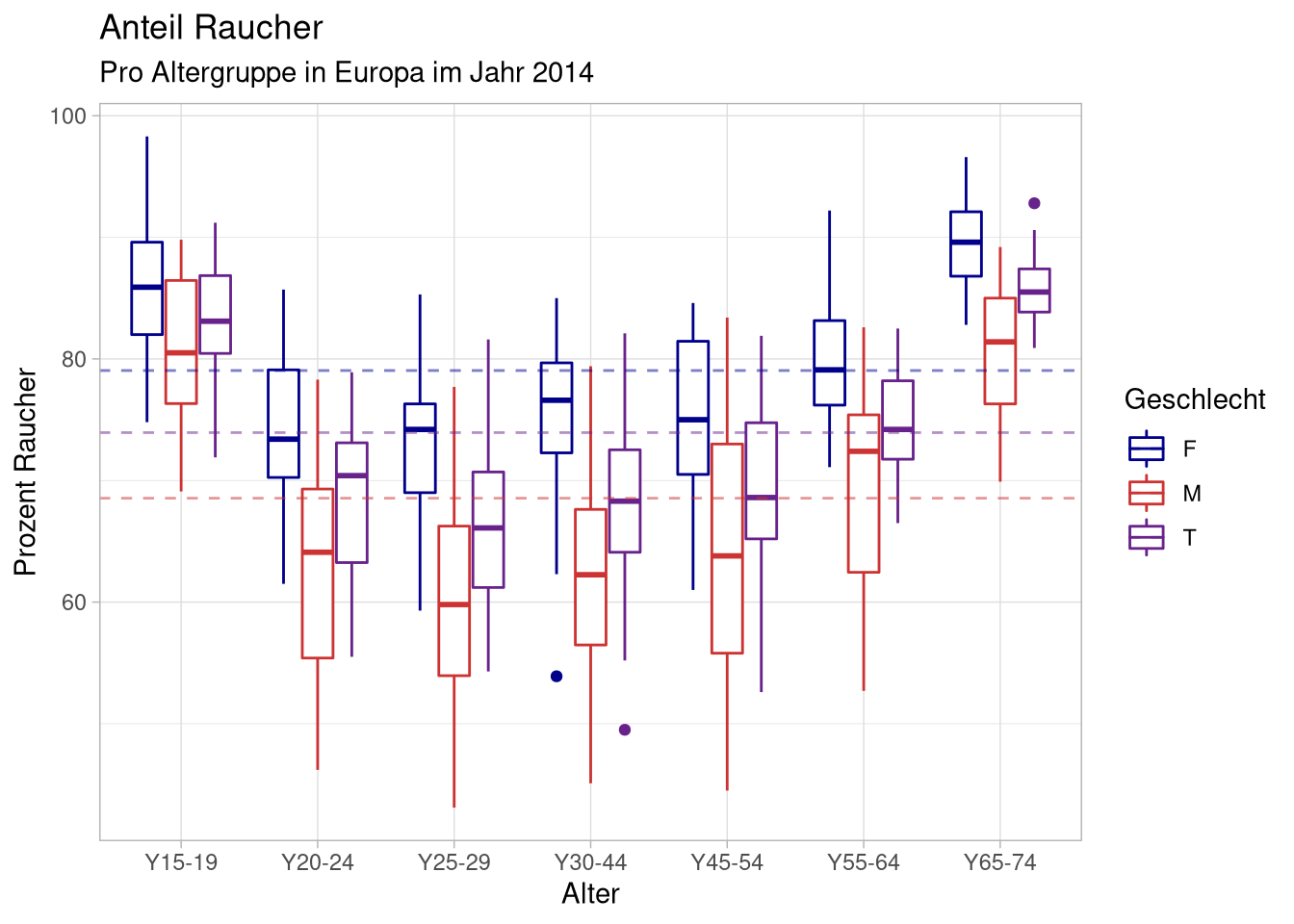 Wir können auch komplexere Informationen nachträglich hinzufügen, so zum Beispiel wenn wir die jeweiligen Mittelwerte als Punkte und die SEMs als Fehlerbalken hinzufügen. Wir fangen wieder damit an, einen Datensatz zu erstellen:
Wir können auch komplexere Informationen nachträglich hinzufügen, so zum Beispiel wenn wir die jeweiligen Mittelwerte als Punkte und die SEMs als Fehlerbalken hinzufügen. Wir fangen wieder damit an, einen Datensatz zu erstellen:
mean_sem_per_box <- df %>%
group_by(sex, age) %>%
summarise(sem = sqrt(var(percentage,na.rm = T) / length(percentage)),
percentage = mean(percentage,na.rm = T),
upper = percentage + sem,
lower = percentage - sem)
mean_sem_per_box## # A tibble: 21 × 6
## # Groups: sex [3]
## sex age sem percentage upper lower
## <chr> <chr> <dbl> <dbl> <dbl> <dbl>
## 1 F Y15-19 1.04 85.8 86.8 84.7
## 2 F Y20-24 1.16 74.2 75.4 73.1
## 3 F Y25-29 1.07 73.7 74.7 72.6
## 4 F Y30-44 1.27 74.7 75.9 73.4
## 5 F Y45-54 1.22 74.8 76.0 73.6
## 6 F Y55-64 0.952 79.7 80.7 78.7
## 7 F Y65-74 0.678 89.8 90.4 89.1
## 8 M Y15-19 1.03 80.8 81.9 79.8
## 9 M Y20-24 1.53 62.8 64.3 61.3
## 10 M Y25-29 1.75 59.3 61.0 57.5
## # … with 11 more rowsDiesen Datensatz können wir dann wieder zu dem Plot hinzufügen:
df %>%
ggplot(aes(x = age,
y = percentage,
color = sex)) +
geom_boxplot() +
scale_color_manual(values = c(F = 'darkblue',
M = 'brown3',
T = 'darkorchid4')) +
labs(x = 'Alter',
y = 'Prozent Raucher',
color = 'Geschlecht',
title = 'Anteil Raucher',
subtitle = 'Pro Altergruppe in Europa im Jahr 2014') +
geom_hline(data = mean_per_sex,
mapping = aes(yintercept = percentage,
color = sex),
alpha = .5,
lty = 2) +
geom_point(data = mean_sem_per_box) +
geom_errorbar(data = mean_sem_per_box,
aes(ymin = lower,
ymax = upper))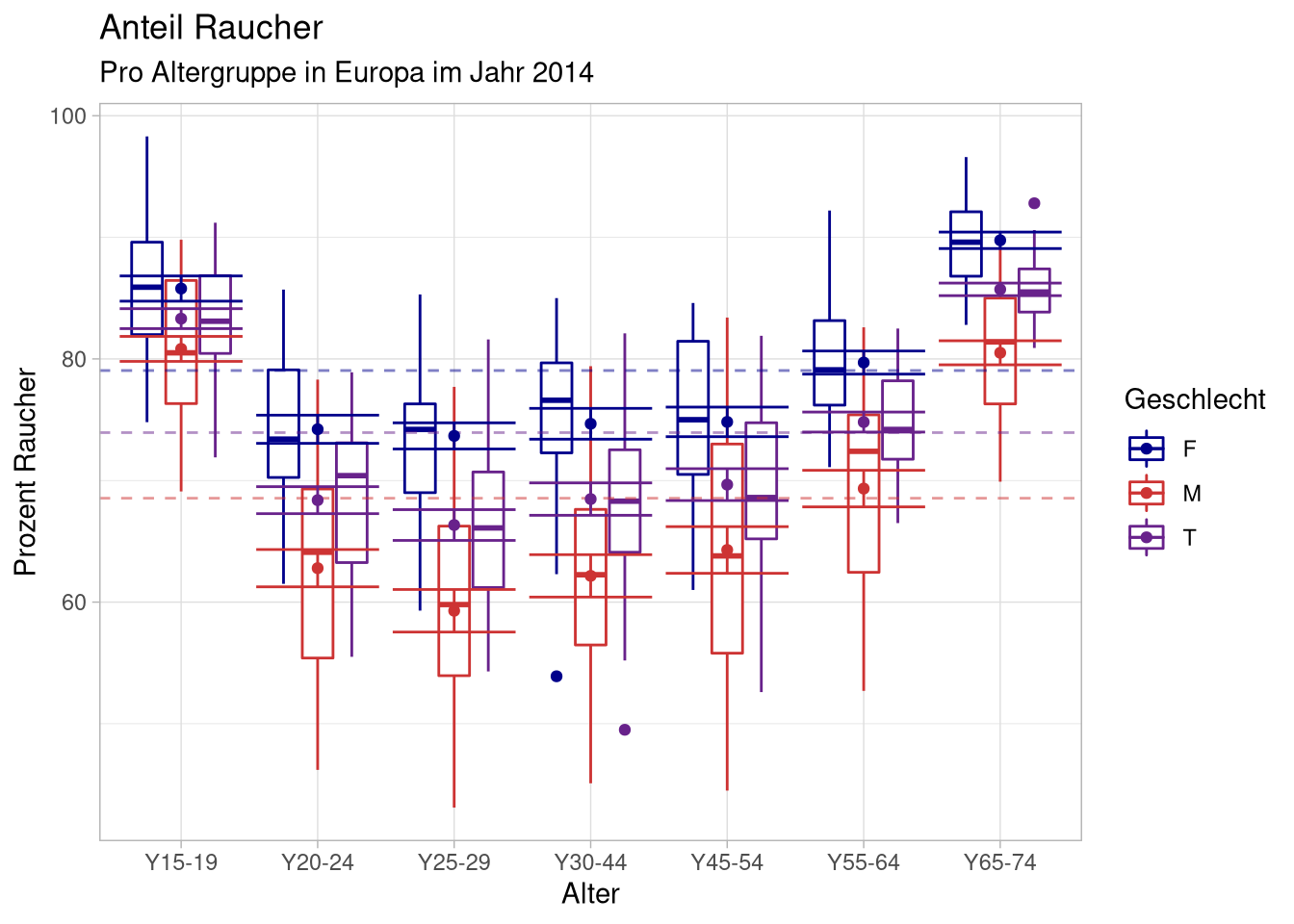 Nicht wirklich optimal, aber in der Richtung sinnvoll. Als ersten Optimierungsschritt benutzen wir wieder das
Nicht wirklich optimal, aber in der Richtung sinnvoll. Als ersten Optimierungsschritt benutzen wir wieder das position-Argument, vie vorher bei den Balkendiagrammen.
df %>%
ggplot(aes(x = age,
y = percentage,
color = sex)) +
geom_boxplot() +
scale_color_manual(values = c(F = 'darkblue',
M = 'brown3',
T = 'darkorchid4')) +
labs(x = 'Alter',
y = 'Prozent Raucher',
color = 'Geschlecht',
title = 'Anteil Raucher',
subtitle = 'Pro Altergruppe in Europa im Jahr 2014') +
geom_hline(data = mean_per_sex,
mapping = aes(yintercept = percentage,
color = sex),
alpha = .5,
lty = 2) +
geom_point(data = mean_sem_per_box,
position = position_dodge()) +
geom_errorbar(data = mean_sem_per_box,
aes(ymin = lower,
ymax = upper),
position = position_dodge())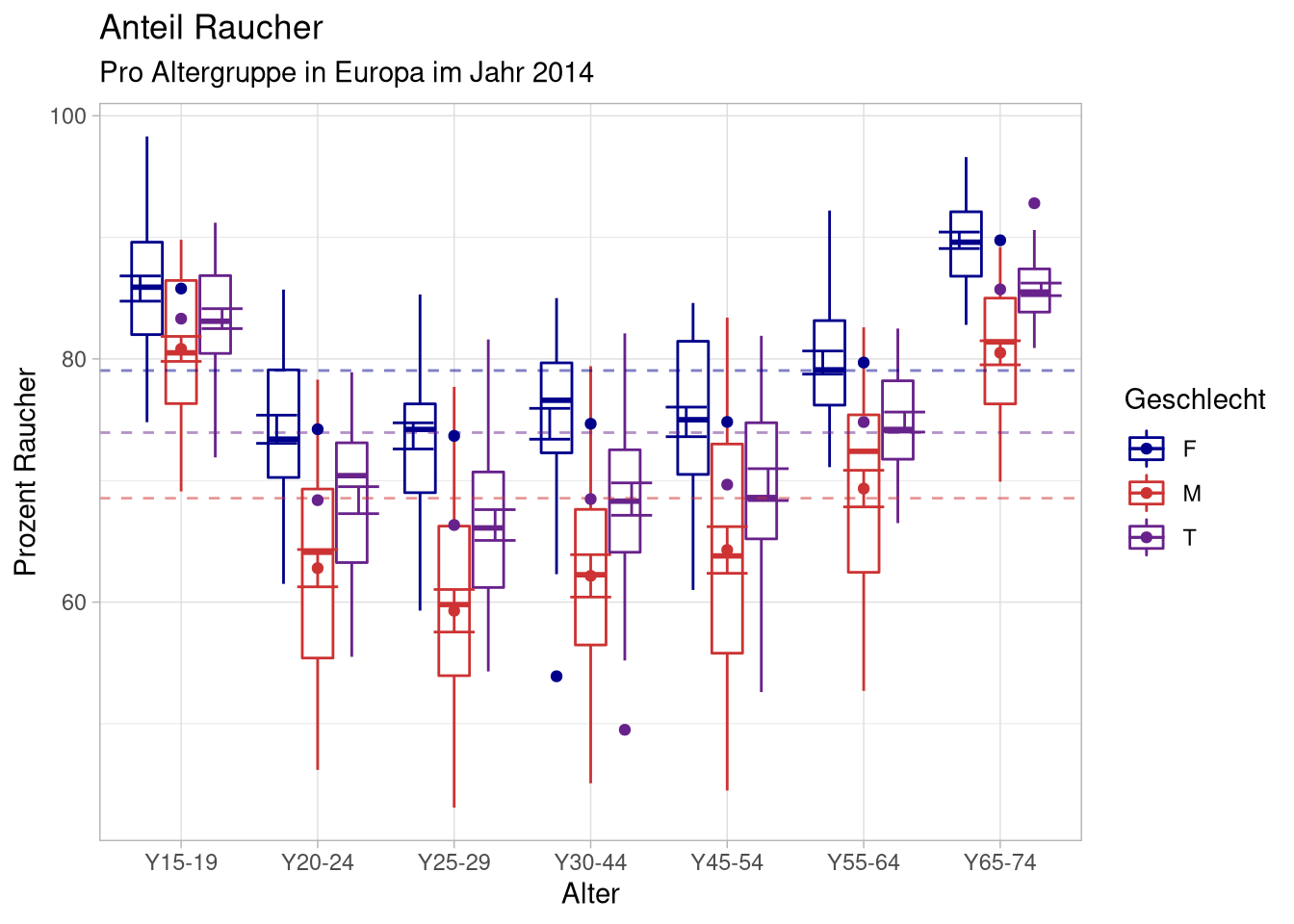
Die position_dodge-Funktion kann ein width-Argument annehmen, das uns die Menge der Verschiebung zu bestimmen erlaubt:
df %>%
ggplot(aes(x = age,
y = percentage,
color = sex)) +
geom_boxplot() +
scale_color_manual(values = c(F = 'darkblue',
M = 'brown3',
T = 'darkorchid4')) +
labs(x = 'Alter',
y = 'Prozent Raucher',
color = 'Geschlecht',
title = 'Anteil Raucher',
subtitle = 'Pro Altergruppe in Europa im Jahr 2014') +
geom_hline(data = mean_per_sex,
mapping = aes(yintercept = percentage,
color = sex),
alpha = .5,
lty = 2) +
geom_point(data = mean_sem_per_box,
position = position_dodge(.75)) +
geom_errorbar(data = mean_sem_per_box,
aes(ymin = lower,
ymax = upper),
position = position_dodge(.75))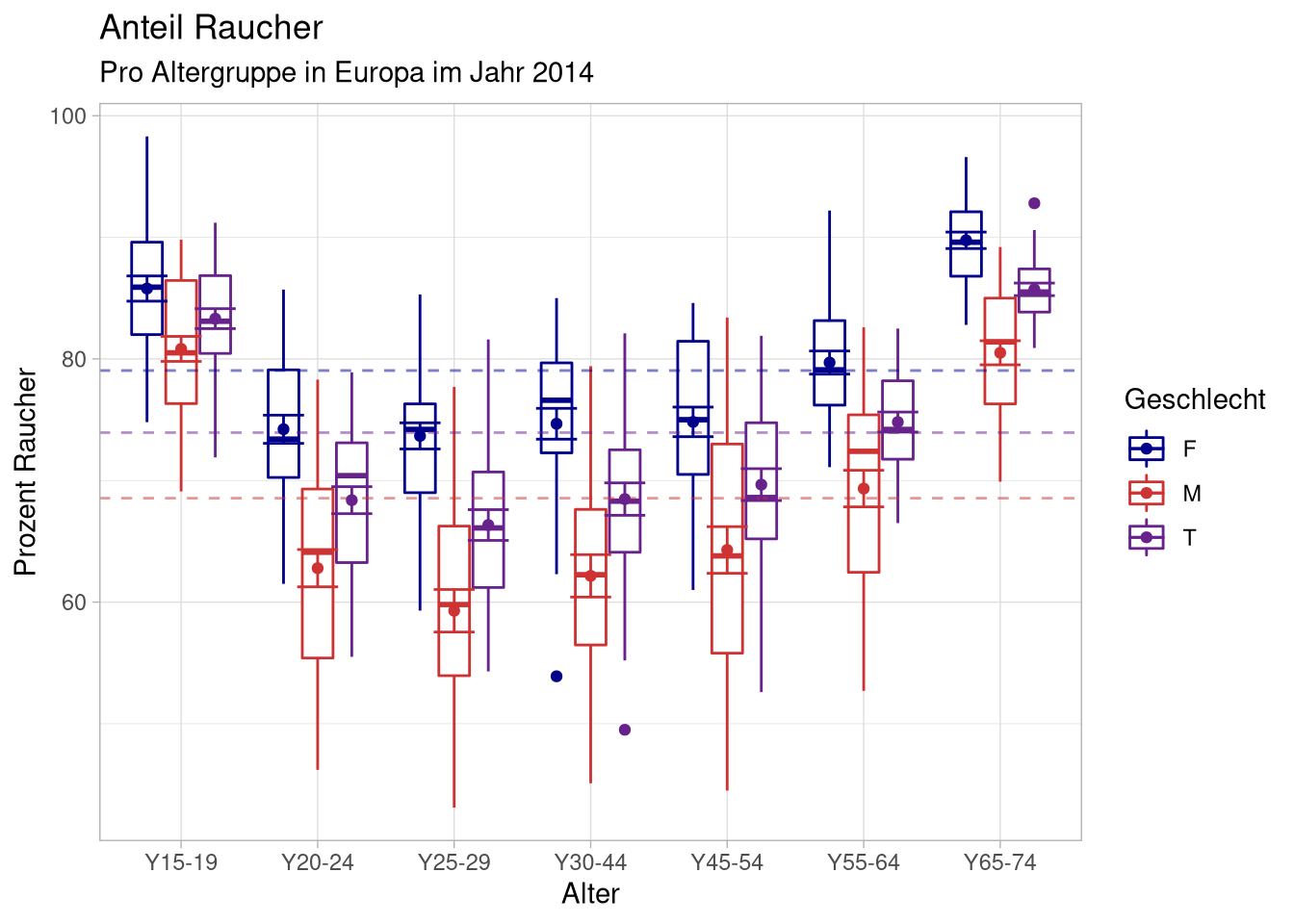
Um das noch ein bisschen zu verbessern, verkleinern wir die Breite der Fehlerbalken und machen die neuen Farben ein wenig transparent um sie abzuheben:
df %>%
ggplot(aes(x = age,
y = percentage,
color = sex)) +
geom_boxplot() +
scale_color_manual(values = c(F = 'darkblue',
M = 'brown3',
T = 'darkorchid4')) +
labs(x = 'Alter',
y = 'Prozent Raucher',
color = 'Geschlecht',
title = 'Anteil Raucher',
subtitle = 'Pro Altergruppe in Europa im Jahr 2014',
caption = "Punkte und Fehlerbalken stellen die jeweiligen Mittelwerte +/- SEM dar, \n die gestrichelten Linien stellen die Mittelwerte pro Geschlechtsgruppe dar.") +
geom_hline(data = mean_per_sex,
mapping = aes(yintercept = percentage,
color = sex),
alpha = .5,
lty = 2) +
geom_point(data = mean_sem_per_box,
position = position_dodge(.75),
alpha = .3) +
geom_errorbar(data = mean_sem_per_box,
aes(ymin = lower,
ymax = upper),
position = position_dodge(.75),
width = .5,
alpha = .3) 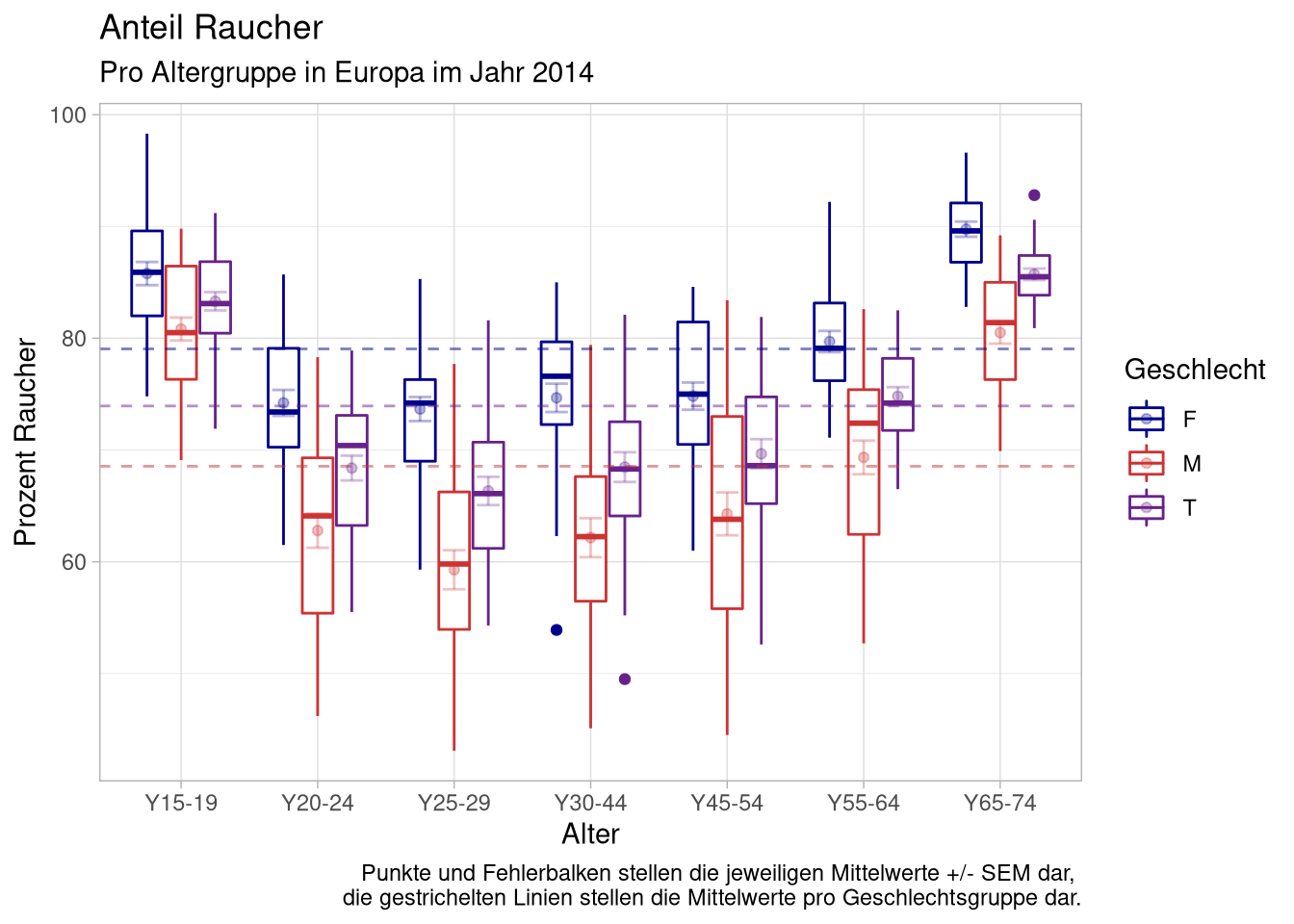
Zusätzliche tweaks
Sortieren von labels
Wir gucken uns den folgenden, von dieser website bezogenen Datensatz an, die die Produktion grüner Kaffeebohnen pro Land in hg/ha beinhaltet:
coffee_df <- read_csv('data/coffee_data.csv')
coffee_df## # A tibble: 77 × 4
## Area Year Unit Value
## <chr> <dbl> <chr> <dbl>
## 1 Angola 2019 hg/ha 3073
## 2 Belize 2019 hg/ha 7273
## 3 Benin 2019 hg/ha 1900
## 4 Bolivia (Plurinational State of) 2019 hg/ha 9742
## 5 Brazil 2019 hg/ha 16504
## 6 Burundi 2019 hg/ha 9136
## 7 Cambodia 2019 hg/ha 8000
## 8 Cameroon 2019 hg/ha 3121
## 9 Central African Republic 2019 hg/ha 2075
## 10 China 2019 hg/ha 30298
## # … with 67 more rowsHektogramm pro Hektar ist ein bisschen ungewöhnlich, deshalb rechnen wir die Angabe in Kilogramm pro Hektar um:
coffee_df <- coffee_df %>%
mutate('kg/ha' = .1 * Value)Diesen Datensatz wollen wir jetzt als Balkendiagramm darstellen:
coffee_df %>%
ggplot(aes(x = Area,
y = `kg/ha`)) +
geom_col(fill = '#59ca19') +
coord_flip()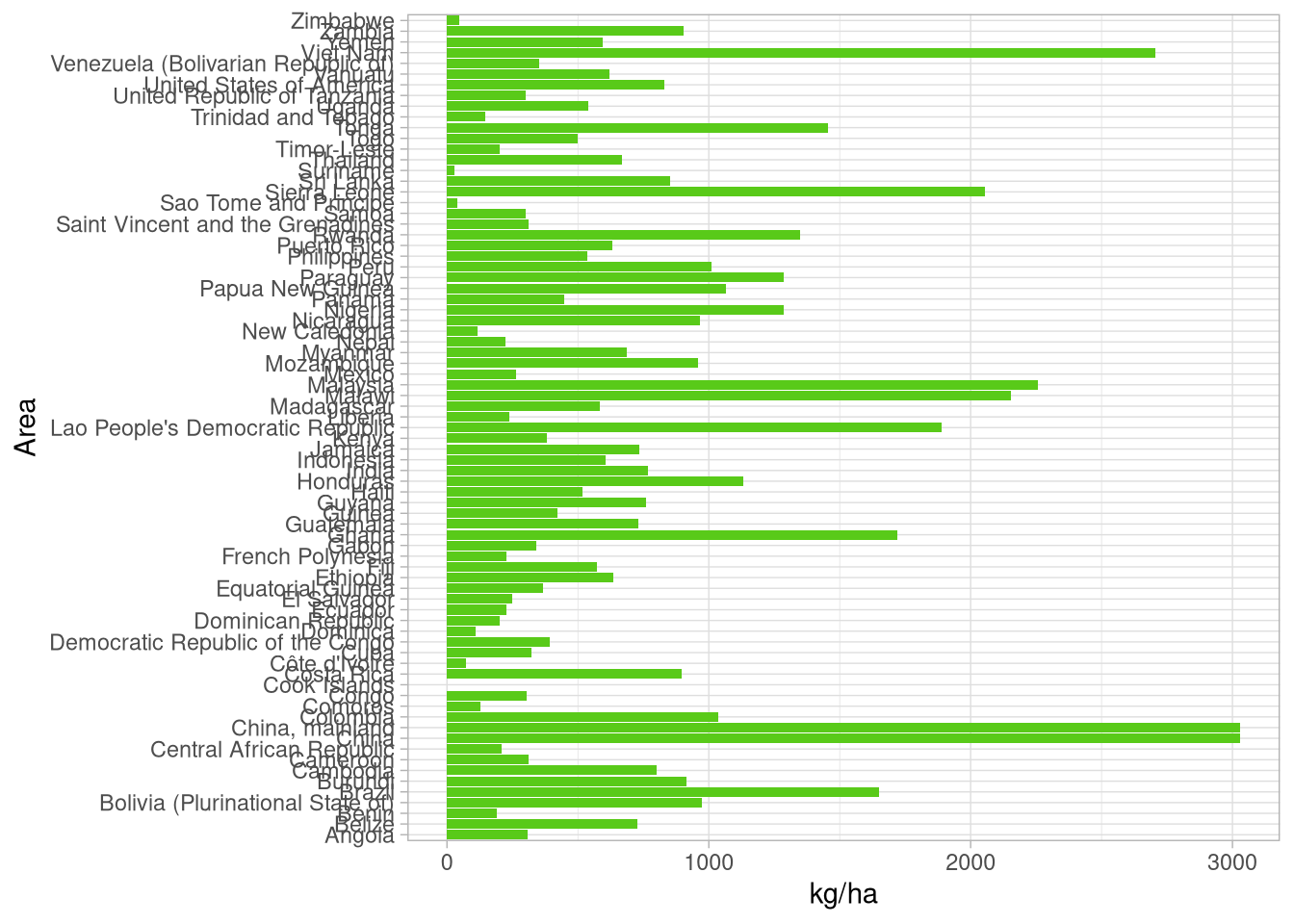
Das sind sehr, sehr viele Einträge. Wir fangen damit an alle Länder unter dem 3. Quartil in eine Kategorie “other” zusammenzufassen und diese Werte dann zu mitteln:
coffee_df %>%
mutate(Area = ifelse(`kg/ha` < quantile(`kg/ha`,probs = .75),
'other (mean)',
Area)) %>%
group_by(Area) %>%
summarise(`kg/ha` = mean(`kg/ha`)) %>%
ggplot(aes(x = Area,
y = `kg/ha`)) +
geom_col(fill = '#59ca19') +
coord_flip()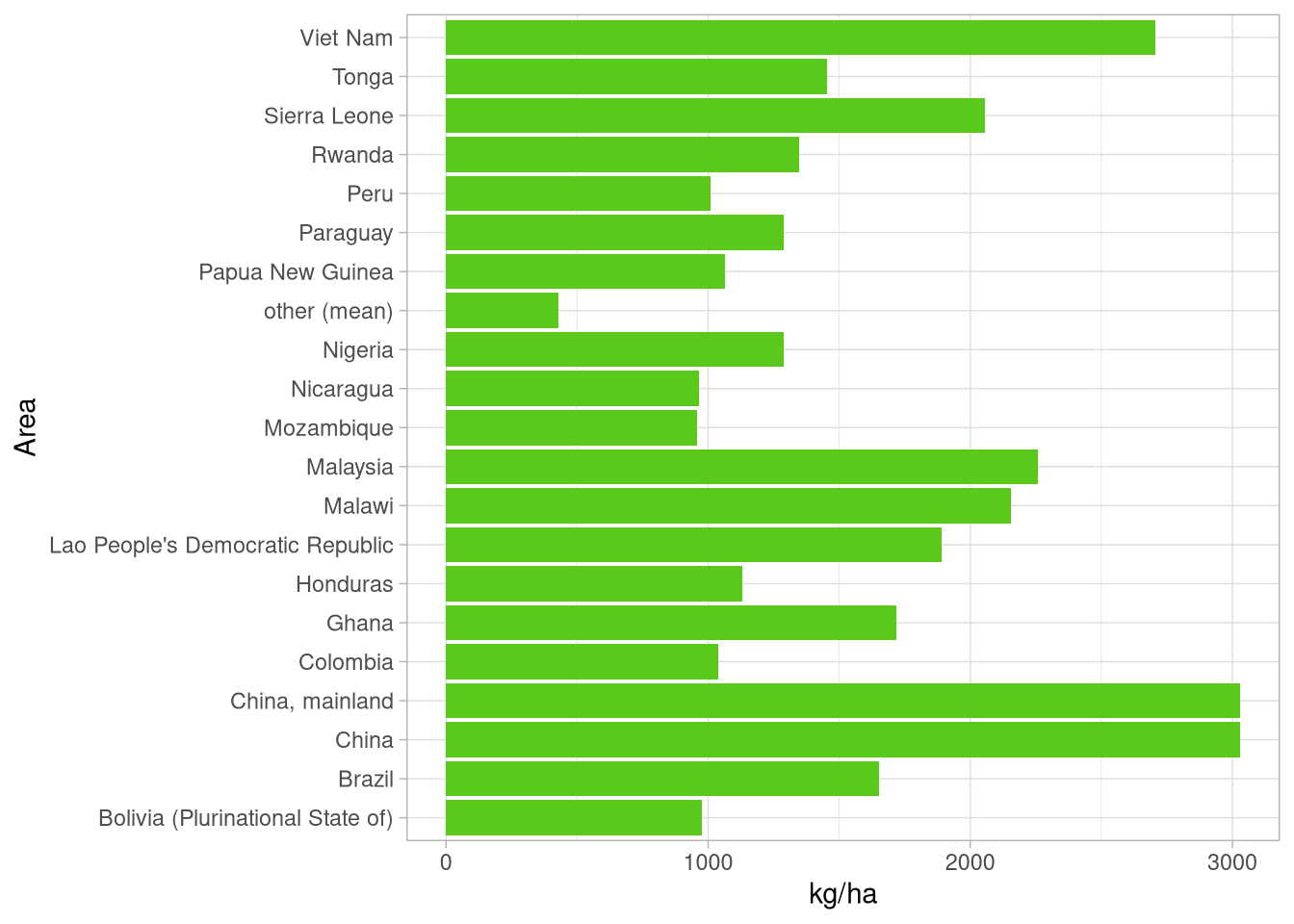
Die y-Achse ist jetzt alphabetisch sortiert. Für einen Länder-Vergleich ist aber eine Ordnung nach Größe vorzuziehen, also machen wir das mal:
coffee_df %>%
mutate(Area = ifelse(`kg/ha` < quantile(`kg/ha`,probs = .75),
'other (mean)',
Area)) %>%
group_by(Area) %>%
summarise(`kg/ha` = mean(`kg/ha`)) %>%
mutate(Area = forcats::fct_reorder(Area, `kg/ha`)) %>%
ggplot(aes(x = Area,
y = `kg/ha`)) +
geom_col(fill = '#59ca19') +
coord_flip() +
labs(x = '',
y = 'Kilogramm pro Hektar',
title = 'Produktionsmenge grünen Kaffees')
facets
Wir betrachten den folgenden Datensatz, der ursprünglich von der open-data Plattform des dwd stammt.
flensburg_kiel_weather <- read_csv("data/flensburg_kiel_weather.csv")
str(flensburg_kiel_weather)## spec_tbl_df [78,283 × 7] (S3: spec_tbl_df/tbl_df/tbl/data.frame)
## $ station : chr [1:78283] "Flensburg" "Flensburg" "Flensburg" "Flensburg" ...
## $ jahr : num [1:78283] 1985 1985 1985 1985 1985 ...
## $ monat : chr [1:78283] "07" "07" "07" "07" ...
## $ tag : chr [1:78283] "01" "02" "03" "04" ...
## $ tagesmittel : num [1:78283] 14.4 13.8 15.6 18 19.2 18.9 14.8 14.2 14.7 15.5 ...
## $ niederschlaege : num [1:78283] 0 0 0 0 0 0 0 7.6 0.2 0 ...
## $ niederschlagsart: chr [1:78283] "kein Niederschlag" "kein Niederschlag" "kein Niederschlag" "kein Niederschlag" ...
## - attr(*, "spec")=
## .. cols(
## .. station = col_character(),
## .. jahr = col_double(),
## .. monat = col_character(),
## .. tag = col_character(),
## .. tagesmittel = col_double(),
## .. niederschlaege = col_double(),
## .. niederschlagsart = col_character()
## .. )
## - attr(*, "problems")=<externalptr>Diesen Datensatz können wir jetzt benutzen um die mittlere Monatstemperatur über die Jahre darzustellen:
flensburg_kiel_weather %>%
group_by(jahr, monat) %>%
summarise(mit_temp = mean(tagesmittel)) %>%
ggplot(aes(x = monat, y = mit_temp)) +
geom_line()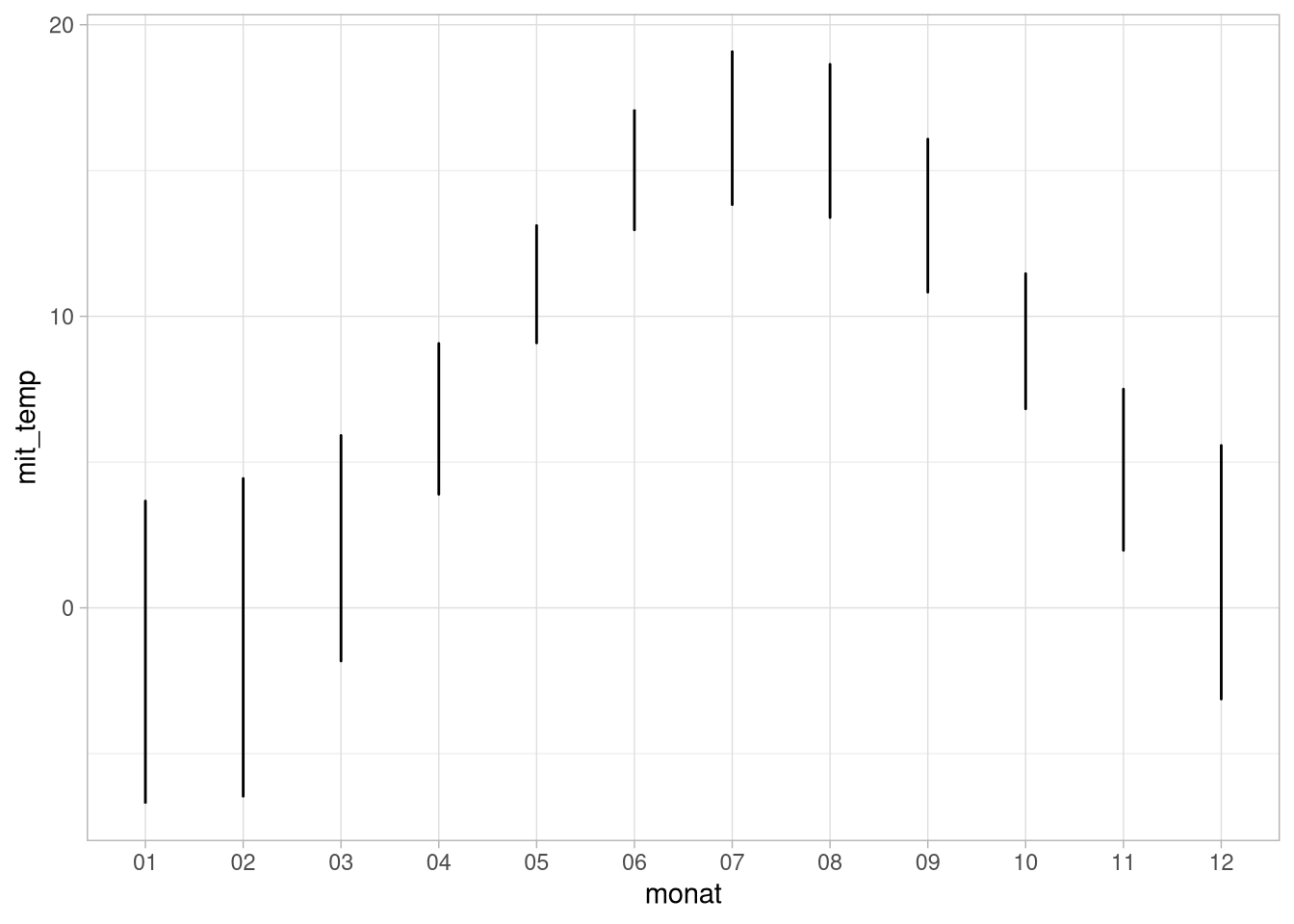 Mit der
Mit der group-aesthetic können wir diesen Graph jetzt per Jahreszahl aufteilen und mit color wieder die Temperatur verdeutlichen:
flensburg_kiel_weather %>%
group_by(jahr, monat) %>%
summarise(mit_temp = mean(tagesmittel)) %>%
ggplot(aes(x = monat,
y = mit_temp,
group = jahr,
color = mit_temp)) +
geom_line() +
scale_color_gradient(low = 'darkblue',
high = 'red') +
labs(y = 'mittlere °C',
color = '°C',
x = 'Monat')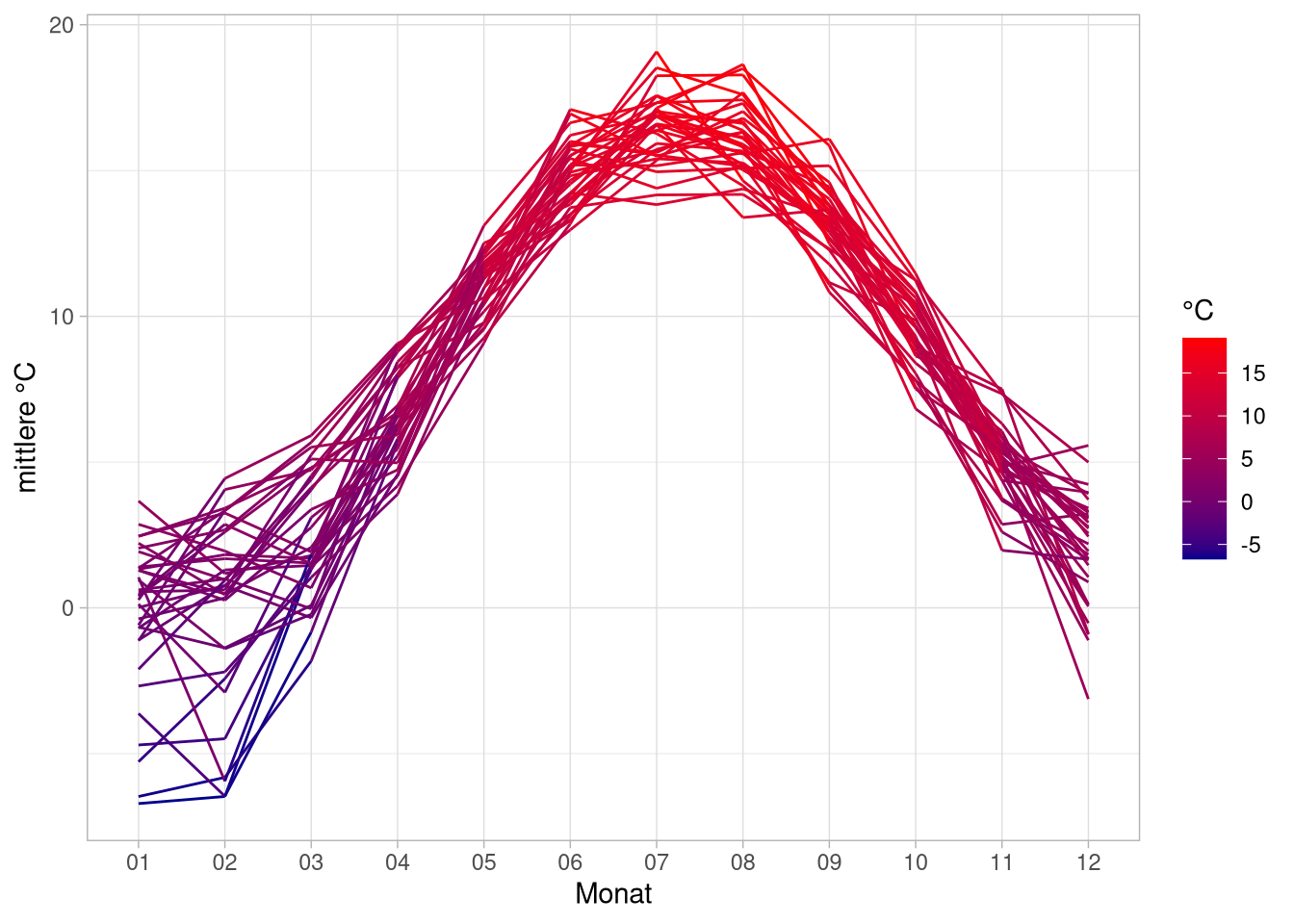 Das ist zwar ganz hübsch, aber nicht alle Informationen, die wir darstellen können.
Im Datensatz sind noch Angaben zu den Messstationen, die wir auch gern unterscheiden würden.
Das ist zwar ganz hübsch, aber nicht alle Informationen, die wir darstellen können.
Im Datensatz sind noch Angaben zu den Messstationen, die wir auch gern unterscheiden würden.
facets können uns helfen, wir können so die einzelnen Stationen in Unterplots darstellen:
flensburg_kiel_weather %>%
group_by(jahr, monat, station) %>%
summarise(mit_temp = mean(tagesmittel)) %>%
ggplot(aes(x = monat,
y = mit_temp,
group = jahr,
color = mit_temp)) +
geom_line() +
scale_color_gradient(low = 'darkblue',
high = 'red') +
labs(y = 'mittlere °C',
color = '°C',
x = 'Monat') +
facet_wrap(~station)
Auf diesen Graphen können wir noch Zusatzinformationen legen, so zum Beipiel einen layer, der die mittleren Monatstemperaturen über alle Jahre pro Station darstellt:
means_per_station <- flensburg_kiel_weather %>%
group_by(monat, station) %>%
summarise(mit_temp = mean(tagesmittel,na.rm = T),
jahr = 'mean')
flensburg_kiel_weather %>%
group_by(jahr, monat, station) %>%
summarise(mit_temp = mean(tagesmittel)) %>%
ggplot(aes(x = monat,
y = mit_temp,
group = jahr,
color = mit_temp)) +
geom_line(alpha = .1) +
geom_line(data = means_per_station) +
scale_color_gradient(low = 'darkblue',
high = 'red') +
labs(y = 'mittlere °C',
color = '°C',
x = 'Monat') +
facet_wrap(~station)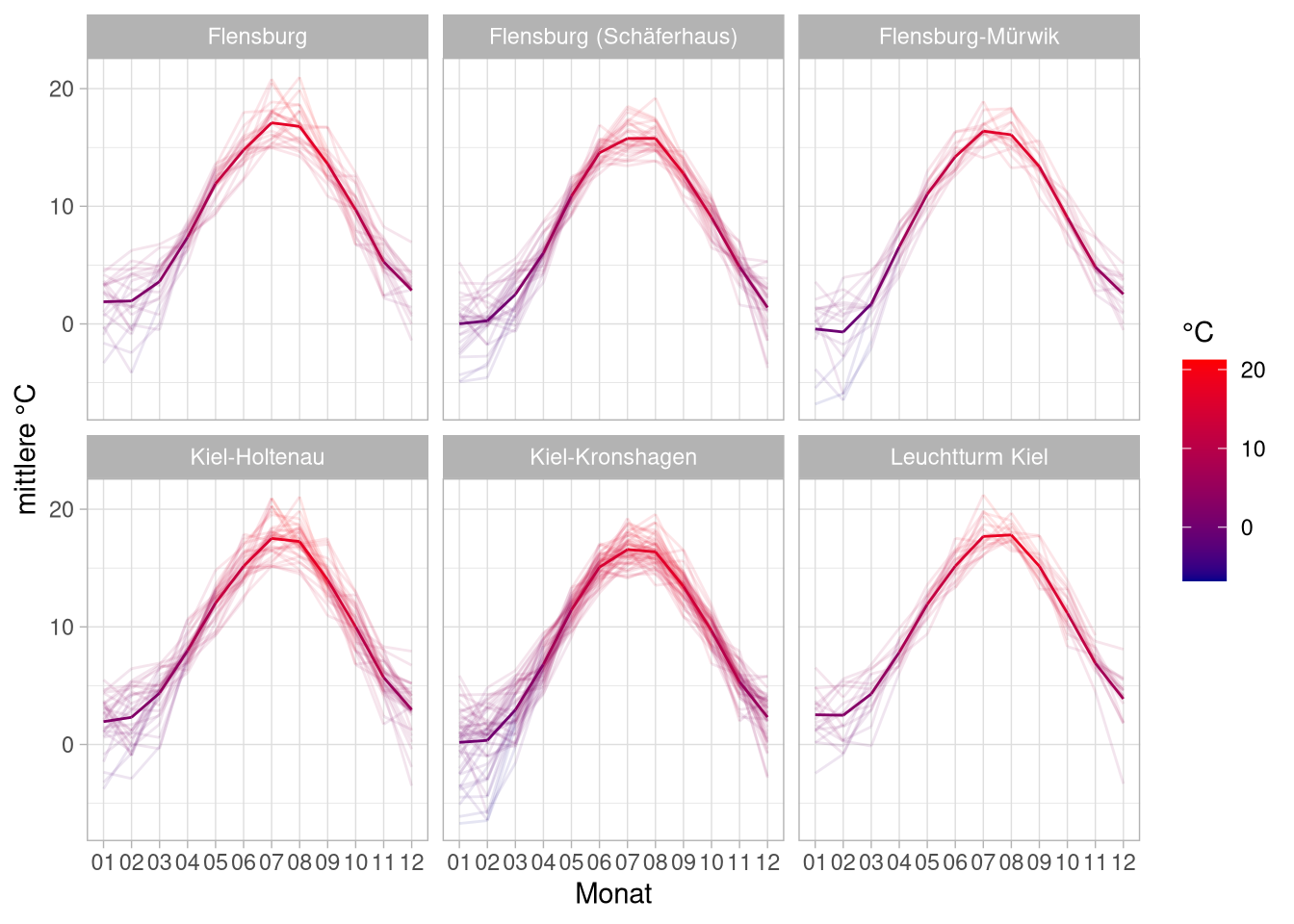
Mit einem Layer nach dem facet-wrap kann man auch einen Wert für alle hinzufügen. So könnten wir uns überlegen, in jeden Plot noch einen Mittelwertsverlauf für alle Stationen aufnehmen zu wollen:
means_per_station <- flensburg_kiel_weather %>%
group_by(monat, station) %>%
summarise(mit_temp = mean(tagesmittel,na.rm = T),
jahr = 'mean')
means_for_all <- flensburg_kiel_weather %>%
group_by(monat) %>%
summarise(mit_temp = mean(tagesmittel,na.rm = T),
jahr = 'mean')
flensburg_kiel_weather %>%
group_by(jahr, monat, station) %>%
summarise(mit_temp = mean(tagesmittel)) %>%
ggplot(aes(x = monat,
y = mit_temp,
group = jahr,
color = mit_temp)) +
geom_line(alpha = .1) +
geom_line(data = means_per_station) +
scale_color_gradient(low = 'darkblue',
high = 'red') +
labs(y = 'mittlere °C',
color = '°C',
x = 'Monat') +
facet_wrap(~station)+
geom_line(data = means_for_all,
color = 'green')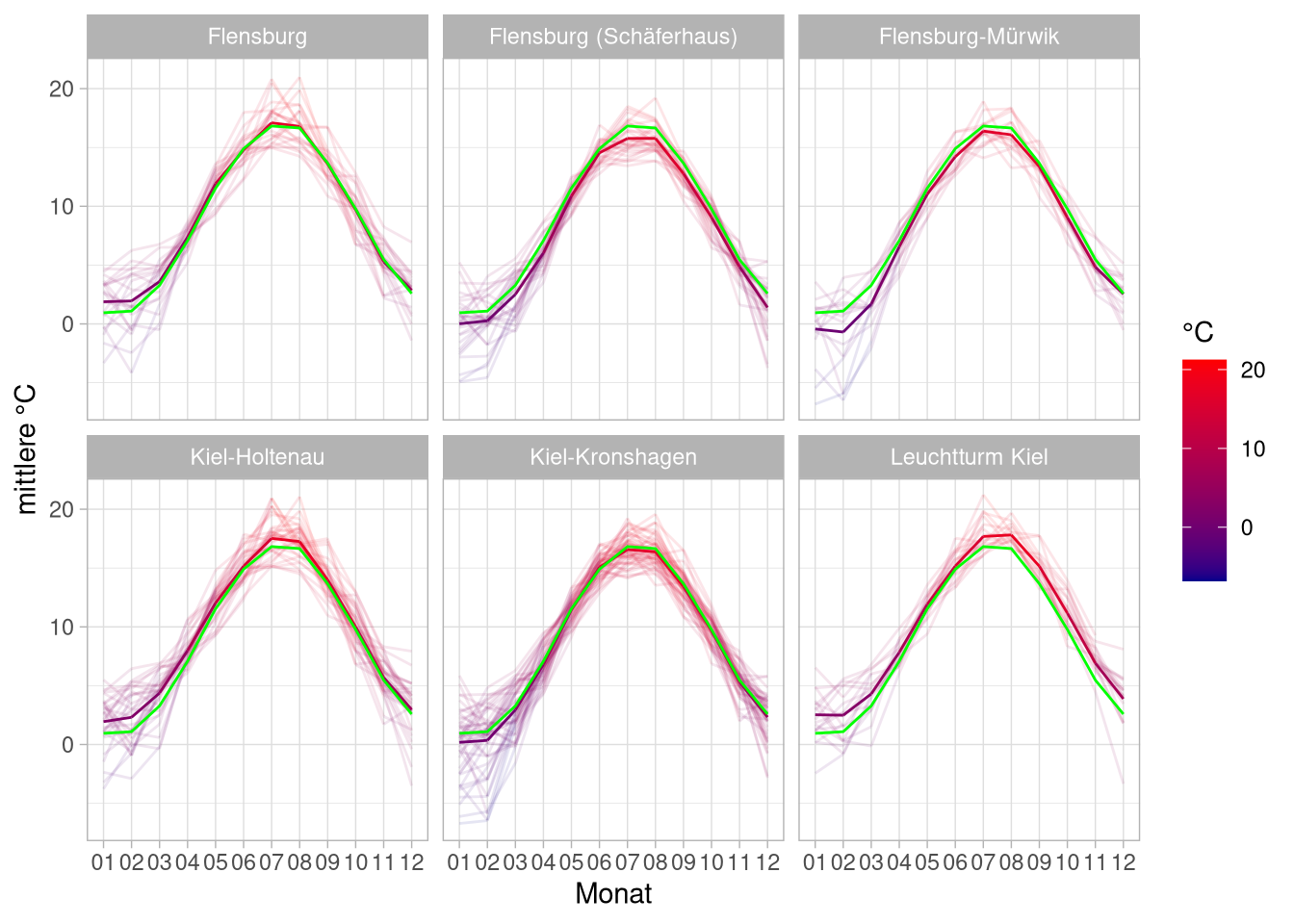
Deskripitv-statische Layer
In dem Datensatz von eben waren auch Werte zur Niederschlagsmenge. Wir könntn uns jetzt die Frage stellen, ob es einen Zusammenhang zwischen der Art und Menge des Niederschlags und der Temperatur gibt.
Dafür erstellen wir zuerst einen Scatterplot, der die Niederschlagsmenge gegen die Temperatur abträgt:
flensburg_kiel_weather %>%
ggplot(aes(x = niederschlaege,
y = tagesmittel)) +
geom_point() 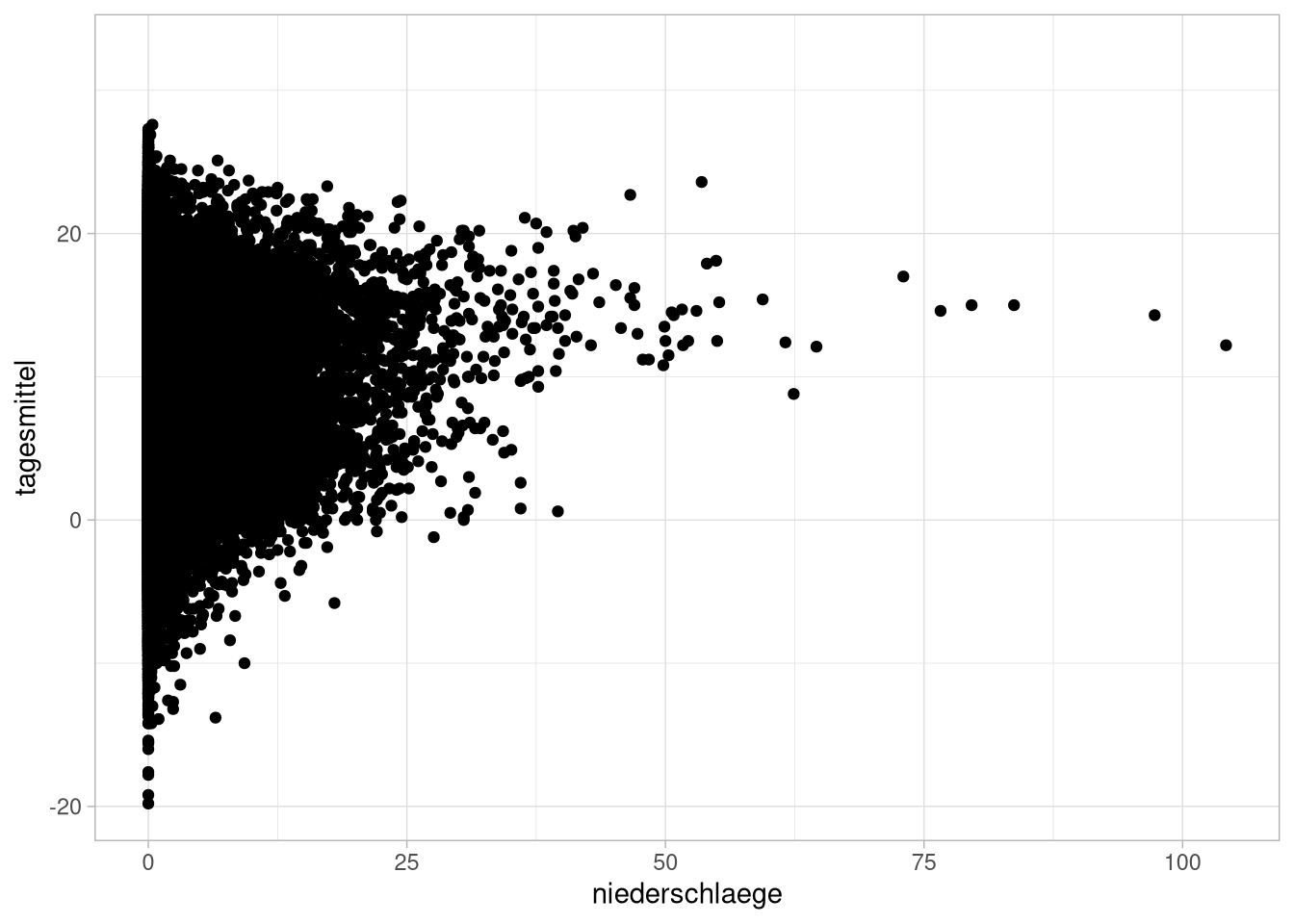
Das ist ein bisschen unübersichtlich, wir aggregieren mal pro Niederschlagsart, Jahr und Station:
flensburg_kiel_weather %>%
group_by(niederschlagsart, jahr, station) %>%
summarise(across(where(is.numeric),mean)) %>%
ggplot(aes(x = niederschlaege,
y = tagesmittel)) +
geom_point()
Jetzt könnten wir die Hypothese haben, dass es eine Korrelation zwischen Temperatur und Niederschlagsmenge gibt. ggplot2 stellt mit der geom_smooth-Funktion eine einfache Möglichkeit dar, eine lineare Regression auf die Daten zu fitten. Wir setzen hier die Argumente method = 'lm' um anzugeben, dass wir ein lineares Modell fitten wollen und das Argument se = F, weil wir keine Fehlerbereiche anzeigen wollen:
flensburg_kiel_weather %>%
group_by(niederschlagsart, jahr, station) %>%
summarise(across(where(is.numeric),mean)) %>%
ggplot(aes(x = niederschlaege,
y = tagesmittel)) +
geom_point() +
geom_smooth(method = 'lm',
se = F)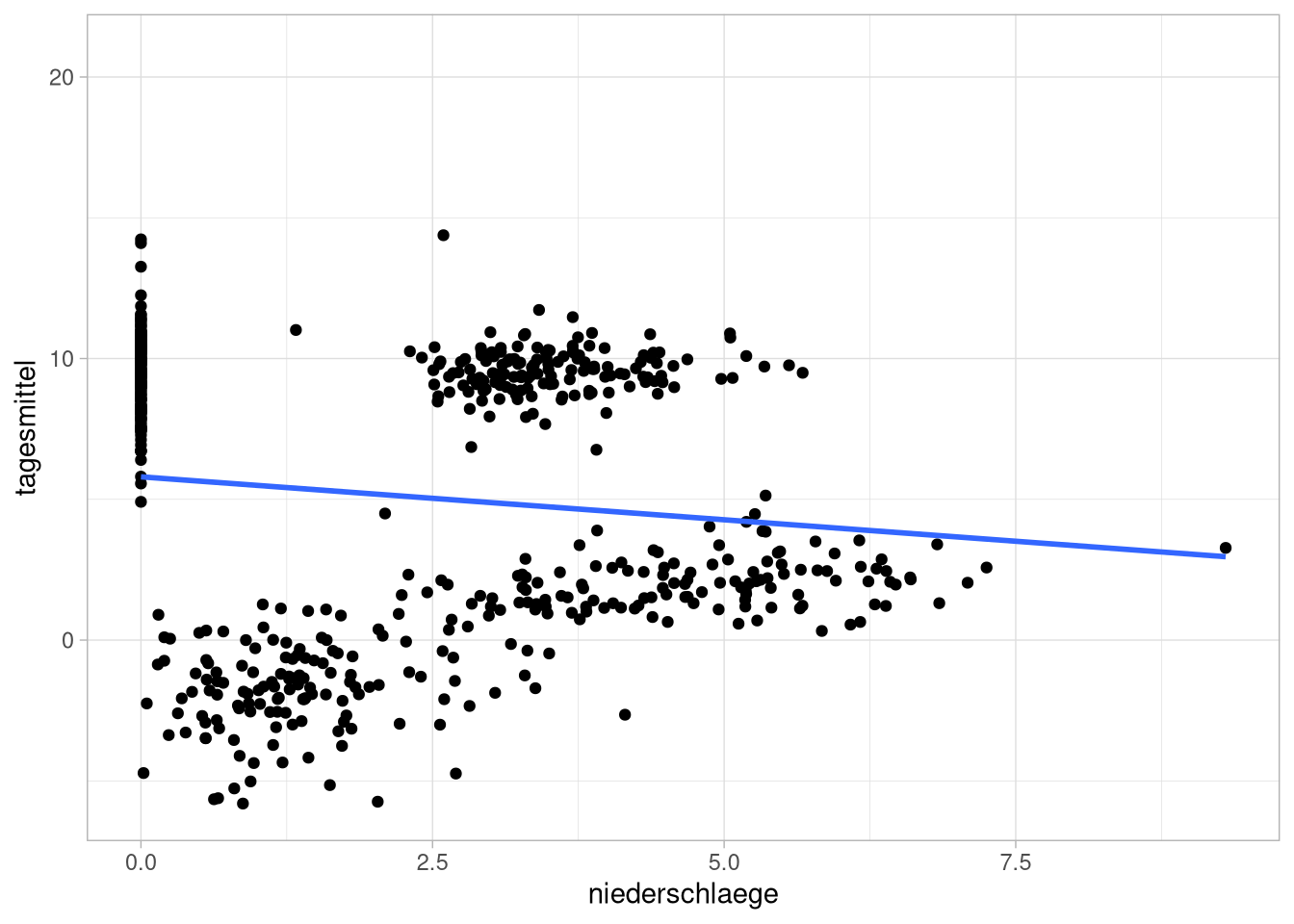
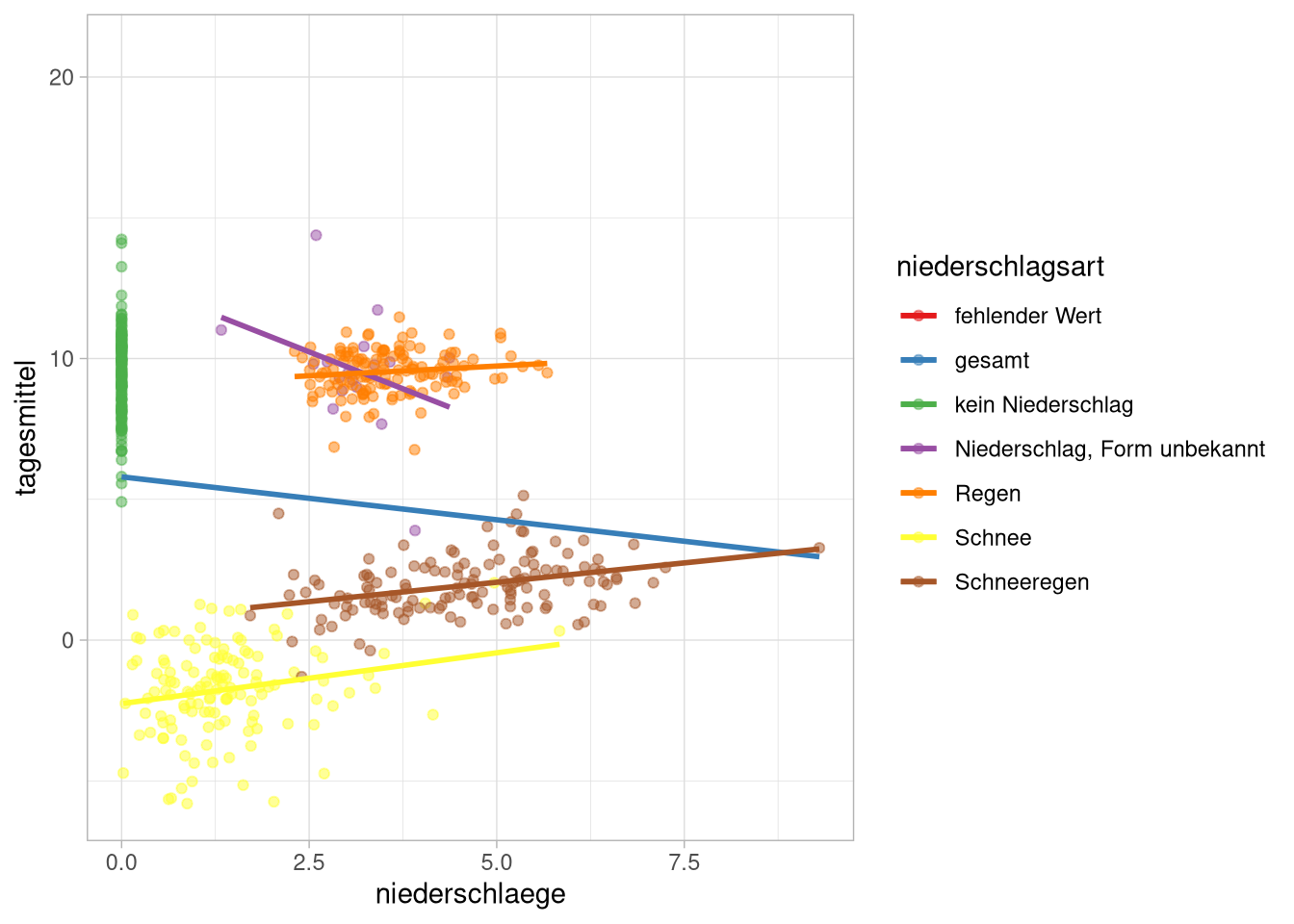
Das ist ein bisschen unbefriedigend. Wenn wir die Niederschlagsart als Gruppierung hinzufügen wird’s aber ein bisschen schicker:
flensburg_kiel_weather %>%
group_by(niederschlagsart, jahr, station) %>%
summarise(across(where(is.numeric),mean)) %>%
ggplot(aes(x = niederschlaege,
y = tagesmittel)) +
geom_point(aes(color = niederschlagsart),
alpha = .5) +
geom_smooth(method = 'lm',
se = F,
aes(color = 'gesamt')) +
geom_smooth(method = 'lm',
se = F,
aes(color = niederschlagsart)) +
scale_color_brewer(palette = 'Set1')